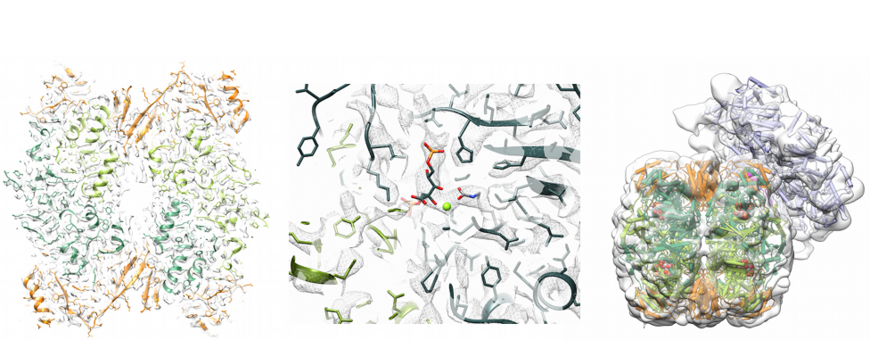
Rubisco Assemblierung und Rubisco Aktivase
Ribulose-1,5-Bisphosphatcarboxylase (Rubisco) vermittelt die Fixierung von Kohlendioxid aus der Atmosphäre in organischen Kohlenstoff während der Photosynthese. Diese Reaktion ist direkt oder indirekt für die Produktion der gesamten Biomasse auf der Erde verantwortlich. Die häufigste Form von Rubisco (Form 1) besteht aus acht großen (RbcL) und acht kleinen (RbcS) Untereinheiten, die einen hexadekamerischen zylindrischen Komplex von 550 kDa bilden. Die RbcL-Untereinheit ist ein klassisches Beispiel für ein Protein, das für die Faltung unbedingt die Unterstützung von Chaperoninen (GroEL in Bakterien) benötigt. Die spontane Montage ist sehr ineffizient. In grüner Form 1 wird die Effizienz der Rubisco-Baugruppe durch das Vorhandensein mehrerer Chaperone wie RbcX, Raf1 und Raf2 weiter verbessert. Form 1 Rubisco ist von photosynthetischen Bakterien bis hin zu Algen und Pflanzen hoch konserviert. Trotz seiner Schlüsselrolle ist Rubisco ein erstaunlich ineffizientes Enzym und seine mehrstufige Reaktion der Carboxylierung ist fehleranfällig, wodurch das aktive Zentrum blockiert wird. Die Reaktivierung des inhibierten Rubisco wird durch das AAA+ Protein Rubisco Activase (Rca) vermittelt.
In Zusammenarbeit mit der Gruppe von Manajit Hayer-Hartl (MPI Martinsried) haben wir kürzlich die Strukturen von Arabidopsis thaliana Raf1 sowie verschiedener Rcas vom grünen und roten Typ mithilfe einer Kombination aus Röntgenkristallographie und Elektronenmikroskopie aufgeklärt.
Arabidopsis thaliana Raf1 (Pflanze; grüner Typ) ist ein Dimer von ~45 kDa Protomeren, die aus einer a-helikalen Domäne und einer Dimerisierungsdomäne bestehen. Ein Komplex aus RbcL8 und vier Raf1-Dimeren bildet das Endzustandsintermediat der Raf1-vermittelten Rubisco-Assemblierung. Unsere Analyse zeigte, dass jedes Raf1-Dimer eine RbcL2 Einheit umfasst. Raf1 hat dabei eine ähnliche Funktion wie der Assemblierungsfaktor RbcX bei der Stabilisierung der RbcL2-Einheit, verwendet jedoch unterschiedliche Interaktionsstellen auf RbcL. Eukaryotisches Rca wird durch das intrazelluläre ATP/ADP-Verhältnis reguliert und liegt in vitro in einem dynamischen Gleichgewicht oligomerer Zustände vor. Die wichtigste aktive Form ist ein ringförmiges Hexamer mit einer klassischen AAA+ Proteindomänenarchitektur.
Nicotiana tabacum Rca (Pflanze; grüner Typ) bildet eine helikale Untereinheit im Kristall und eine hexamere Ringanordnung in der EM-Analyse. Die 7,5 kDa N-terminale Domäne von NtRca befindet sich oben auf dem Hexamer und vermittelt vermutlich die Interaktion mit Rubisco. Der Mechanismus der Rubisco-Remodellierung durch eukaryotische Rca ist jedoch nicht bekannt. Unsere Studie zeigte, dass Mutationen in den Porenschleifen des Hexamers zu einer Verringerung der Rubisco-Remodellierungsaktivität und/oder ATPase-Aktivität führen.
Die strukturelle und funktionelle Analyse von prokaryotischen Rhodobacter sphaeroides Rca (bakteriell; roter Typ) lieferte kritische Einblicke in den Mechanismus der Rubisco-Remodellierung. Interessanterweise bildet sich die aktive hexamere Form von Rca nur in Gegenwart von ATP und Ribulose-Bisphosphat (RuBP), dem Substrat seines Zielenzyms Rubisco. Die Hexamerporen sind mit kanonischen, mit dem Substrat wechselwirkenden Rückständen ausgekleidet, die für die Umgestaltung von Rubisco entscheidend sind. Darüber hinaus erfordert die Reaktivierung von Rubisco eine intakte C-terminale Sequenz von RbcL. Unsere Kryo-EM-Rekonstruktion des Rhodobacter sphaeroides Rca: Rubisco-Komplexes ergab, dass Rca mit seiner oberen Oberfläche an Rubisco andockt und den C-terminalen Schwanz von RbcL vorübergehend in die zentrale Pore zieht, um das Öffnen der Tasche des aktiven Zentrums und die Freisetzung des Inhibitors zu erleichtern Zuckerphosphat.
Veröffentlichungen:
Bhat, J. Y., G. Miličić, G. Thieulin-Pardo, A. Bracher, A. Maxwell, S. Ciniawsky, O. Mueller-Cajar, J. R. Engen, F. U. Hartl, P. Wendler, M. Hayer-Hartl (2017). Molecular Surgery: Enzyme Repair by the AAA+ Chaperone Rubisco Activase. Mol Cell (in press) http://www.cell.com/molecular-cell/fulltext/S1097-2765(17)30498-7
Hauser, T., J. Y. Bhat, G. Miličić, P. Wendler, F. U. Hartl, A. Bracher, M. Hayer-Hartl (2015). Structure and mechanism of the Rubisco assembly chaperone Raf1. Nat Struc Mol Biol. 22(9):720-8
https://www.nature.com/nsmb/journal/v22/n9/full/nsmb.3062.html
Stotz M, O. Mueller-Cajar, S. Ciniawsky, P. Wendler, U. Hartl, A. Bracher, M. Hayer-Hartl (2011). Structure of green-type rubisco activase from tobacco. Nat Struc Mol Biol 18(12):1366-70.
http://www.nature.com/nsmb/journal/v18/n12/full/nsmb.2171.html
Mueller-Cajar, O., M. Stotz, P. Wendler, F.U. Hartl, A. Bracher, M. Hayer-Hartl (2011). Structure and function of the AAA+ protein CbbX, a red-type Rubisco activase. Nature 479(7372):194-9.
https://www.nature.com/nature/journal/v479/n7372/full/nature10568.html